Pesquisadores do Instituto Oswaldo Cruz (IOC/Fiocruz) traçaram a evolução e dispersĂŁo de um dos perfis genĂ©ticos do vĂrus da hepatite B nas AmĂ©ricas, responsável por casos de desenvolvimento de câncer de fĂgado. Conhecido como genĂłtipo D, este perfil tambĂ©m está relacionado a baixas respostas ao tratamento pelo interferon, um dos principais medicamentos usados no combate Ă infecção. Segundo estimativas da Organização Mundial da SaĂşde (OMS), há, ao menos, 257 milhões de portadores crĂ´nicos da hepatite B em todo o mundo.
“Amplamente distribuĂdo no planeta, o vĂrus da hepatite B Ă© dividido em pelo menos dez genĂłtipos, que sĂŁo identificados por letras de A a J. Com distribuição geográfica distinta, esses perfis genĂ©ticos apresentam diferenças significativas na progressĂŁo da doença, resposta Ă terapia antiviral e desfechos clĂnicos”, explicou Natalia Motta de Araujo, pesquisadora do LaboratĂłrio de Virologia Molecular do IOC e coordenadora do estudo.
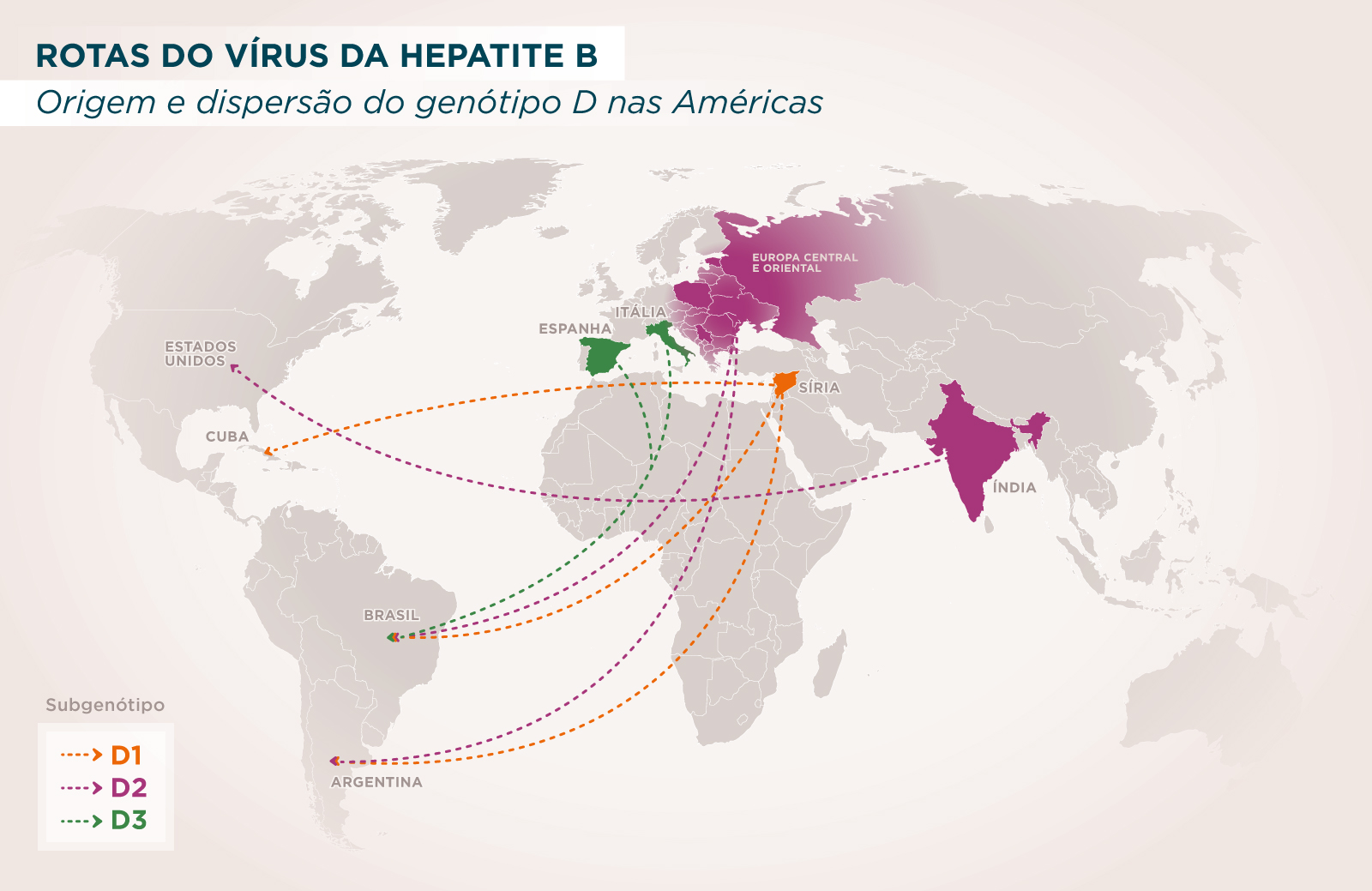
ApĂłs análise de dados do sequenciamento completo do genoma do vĂrus, realizado pelo grupo a partir de amostras brasileiras, e de sequĂŞncias genĂ©ticas de paĂses do continente americano disponĂveis em um banco de dados internacional, os cientistas apontam que as principais rotas de introdução do genĂłtipo D na regiĂŁo estĂŁo relacionadas a movimentos de migração em massa, entre os sĂ©culos XIX e XX, com origem principalmente na SĂria, LĂbano, ĂŤndia e em paĂses da Europa Central e Oriental.
“Conhecer as atividades humanas relacionadas Ă dispersĂŁo de um genĂłtipo Ă© importante para a vigilância em saĂşde, com definições, por exemplo, de estratĂ©gias de prevenção. Ampliar o conhecimento dos genĂłtipos que circulam numa regiĂŁo contribui para o desenvolvimento de estudos acerca de diferenças clĂnicas e de respostas terapĂŞuticas que podem ocorrer devido Ă genĂ©tica do vĂrus”, destacou a pesquisadora.
Os pesquisadores realizaram o sequenciamento completo do genoma de 39 amostras, das cinco regiões do Brasil, de pacientes infectados com o genĂłtipo D do vĂrus da hepatite B. Somaram-se Ă análise 1.135 sequĂŞncias (209 completas e 926 parciais) isoladas no Brasil, Argentina, Canadá, Chile, ColĂ´mbia, Cuba, Haiti, Martinica, MĂ©xico, Estados Unidos e Venezuela.
Com base em análises filogenĂ©ticas – uma espĂ©cie de árvore genealĂłgica, sĂł que do vĂrus -, os pesquisadores estabeleceram um panorama ainda mais detalhado considerando os subgenĂłtipos que circulam no Brasil e nas AmĂ©ricas. Eles descobriram que, dos dez subgenĂłtipos (do genĂłtipo D) existentes, pelo menos cinco circulam no continente americano (D1, D2, D3, D4 e D7). Destes, apenas o D7 nĂŁo foi identificado como circulante no Brasil. O subgenĂłtipo D1 foi o mais prevalente na Argentina e Canadá, assim como o D2 nos Estados Unidos, o D3 no Brasil, e o D4 em Cuba e no Haiti. O que diferencia os subgenĂłtipos sĂŁo caracterĂsticas bem especĂficas.

O sequenciamento de 39 amostras brasileiras, realizado no estudo, equivale a 19% dos genomas completos do genĂłtipo D disponĂveis para consulta no continente americano (Foto: Gutemberg Brito/IOC)
Para estimar a origem e data de introdução dos subgenĂłtipos circulantes nas AmĂ©ricas, os pesquisadores analisaram as sequĂŞncias genĂ©ticas com o uso de ferramentas de bioinformática que consideram principalmente o local de origem e a data de coleta das amostras. A reconstrução espaço-temporal mostrou que o subgenĂłtipo D1 possivelmente tenha origem na SĂria, de onde chegou ao Brasil, Cuba e Argentina em ocasiões diferentes.
Já a análise filogeográfica do subgenĂłtipo D2 sugeriu que o mais provável epicentro tenha sido a Europa Central e Oriental, com destaque para a RĂşssia, incluindo paĂses como EstĂ´nia, PolĂ´nia e SĂ©rvia. As sequĂŞncias genĂ©ticas dos Estados Unidos, quando agrupadas com as indianas, apontaram que pelo menos duas introduções diferentes deste subgenĂłtipo ocorreram no paĂs norte-americano a partir da ĂŤndia. O principal perĂodo de introdução foi estimado entre 1966 e 1978.
O subgenótipo D3 apresentou vias de dispersão complexas, incluindo diferentes regiões geográficas e múltiplas introduções. As análises destacaram que o D3 tenha como origem provável o Sul da Europa (Itália e Espanha) e o Brasil como fonte de dispersão para o continente. O subgenótipo D3 foi encontrado em todas as regiões do Brasil, com as mais altas taxas na Região Sul, que recebeu um fluxo intenso de imigrantes europeus.
A investigação do D4 demonstrou uma maior prevalĂŞncia desse subgenĂłtipo em paĂses americanos onde a população majoritária Ă© de descendentes de africanos. Entretanto, os resultados sobre o local de origem do D4 foram inconclusivos. “Somente nĂŁo foi possĂvel desenhar a árvore filogenĂ©tica do subgenĂłtipo D7, devido Ă sua natureza recombinante e ao nĂşmero limitado de sequĂŞncias disponĂveis”, ponderou a especialista.
Os pesquisadores destacaram que, de forma majoritária, os achados foram compatĂveis com dados histĂłricos e epidemiolĂłgicos, demonstrando assim a utilidade das ferramentas de bioinformática em estudos evolutivos. A migração europeia em massa para as AmĂ©ricas ocorreu do inĂcio do sĂ©culo XIX atĂ© meados do sĂ©culo XX, especialmente para os Estados Unidos, Argentina, Canadá, Brasil, Cuba e Uruguai. Em perĂodo semelhante, milhares de pessoas, principalmente da SĂria, LĂbano, Palestina, Turquia e Egito migraram para Cuba. Em relação ao Brasil, o movimento migratĂłrio de árabes, em especial de paĂses como SĂria e LĂbano, tambĂ©m Ă© documentado a partir da segunda metade do sĂ©culo XIX. Já nos Estados Unidos, um dos maiores grupos de imigrantes tem como origem a ĂŤndia.
O estudo foi realizado como parte do projeto de doutorado da estudante Natália Spitz Toledo Dias, do Programa de PĂłs-graduação Stricto sensu em Biologia Parasitária do IOC. “As análises tambĂ©m permitiram conhecer melhor a velocidade com que o vĂrus evolui e a forma como ele vai se modificando ao longo do tempo”, frisou. “Determinadas mutações sĂŁo importantes do ponto de vista clĂnico, uma vez que podem gerar resistĂŞncia a um medicamento antiviral, falha na resposta Ă vacina ou fazer com que o microrganismo nĂŁo seja reconhecido nos kits de diagnĂłstico, por exemplo”, completou.
A pesquisa, publicada no periĂłdico cientĂfico Plos One, tambĂ©m contou com a contribuição dos pesquisadores do IOC Gonzalo Bello, do LaboratĂłrio de Aids e Imunologia Molecular; Selma Gomes, do LaboratĂłrio de Virologia Molecular; Francisco Mello, do LaboratĂłrio de Hepatites Virais, e Aline Moreira, do LaboratĂłrio de GenĂ´mica Funcional e Bioinformática. Participaram, ainda, especialistas da Universidade Federal de Goiás (UFG) e da Fundação Estadual de Produção e Pesquisa em SaĂşde (FEPPS), do Rio Grande do Sul.

Pesquisadores do Instituto Oswaldo Cruz (IOC/Fiocruz) traçaram a evolução e dispersĂŁo de um dos perfis genĂ©ticos do vĂrus da hepatite B nas AmĂ©ricas, responsável por casos de desenvolvimento de câncer de fĂgado. Conhecido como genĂłtipo D, este perfil tambĂ©m está relacionado a baixas respostas ao tratamento pelo interferon, um dos principais medicamentos usados no combate Ă infecção. Segundo estimativas da Organização Mundial da SaĂşde (OMS), há, ao menos, 257 milhões de portadores crĂ´nicos da hepatite B em todo o mundo.
“Amplamente distribuĂdo no planeta, o vĂrus da hepatite B Ă© dividido em pelo menos dez genĂłtipos, que sĂŁo identificados por letras de A a J. Com distribuição geográfica distinta, esses perfis genĂ©ticos apresentam diferenças significativas na progressĂŁo da doença, resposta Ă terapia antiviral e desfechos clĂnicos”, explicou Natalia Motta de Araujo, pesquisadora do LaboratĂłrio de Virologia Molecular do IOC e coordenadora do estudo.
ApĂłs análise de dados do sequenciamento completo do genoma do vĂrus, realizado pelo grupo a partir de amostras brasileiras, e de sequĂŞncias genĂ©ticas de paĂses do continente americano disponĂveis em um banco de dados internacional, os cientistas apontam que as principais rotas de introdução do genĂłtipo D na regiĂŁo estĂŁo relacionadas a movimentos de migração em massa, entre os sĂ©culos XIX e XX, com origem principalmente na SĂria, LĂbano, ĂŤndia e em paĂses da Europa Central e Oriental.Â
“Conhecer as atividades humanas relacionadas Ă dispersĂŁo de um genĂłtipo Ă© importante para a vigilância em saĂşde, com definições, por exemplo, de estratĂ©gias de prevenção. Ampliar o conhecimento dos genĂłtipos que circulam numa regiĂŁo contribui para o desenvolvimento de estudos acerca de diferenças clĂnicas e de respostas terapĂŞuticas que podem ocorrer devido Ă genĂ©tica do vĂrus”, destacou a pesquisadora.
Os pesquisadores realizaram o sequenciamento completo do genoma de 39 amostras, das cinco regiões do Brasil, de pacientes infectados com o genĂłtipo D do vĂrus da hepatite B. Somaram-se Ă análise 1.135 sequĂŞncias (209 completas e 926 parciais) isoladas no Brasil, Argentina, Canadá, Chile, ColĂ´mbia, Cuba, Haiti, Martinica, MĂ©xico, Estados Unidos e Venezuela.
Com base em análises filogenĂ©ticas – uma espĂ©cie de árvore genealĂłgica, sĂł que do vĂrus -, os pesquisadores estabeleceram um panorama ainda mais detalhado considerando os subgenĂłtipos que circulam no Brasil e nas AmĂ©ricas. Eles descobriram que, dos dez subgenĂłtipos (do genĂłtipo D) existentes, pelo menos cinco circulam no continente americano (D1, D2, D3, D4 e D7). Destes, apenas o D7 nĂŁo foi identificado como circulante no Brasil. O subgenĂłtipo D1 foi o mais prevalente na Argentina e Canadá, assim como o D2 nos Estados Unidos, o D3 no Brasil, e o D4 em Cuba e no Haiti. O que diferencia os subgenĂłtipos sĂŁo caracterĂsticas bem especĂficas.

O sequenciamento de 39 amostras brasileiras, realizado no estudo, equivale a 19% dos genomas completos do genĂłtipo D disponĂveis para consulta no continente americano (Foto: Gutemberg Brito/IOC)
Para estimar a origem e data de introdução dos subgenĂłtipos circulantes nas AmĂ©ricas, os pesquisadores analisaram as sequĂŞncias genĂ©ticas com o uso de ferramentas de bioinformática que consideram principalmente o local de origem e a data de coleta das amostras. A reconstrução espaço-temporal mostrou que o subgenĂłtipo D1 possivelmente tenha origem na SĂria, de onde chegou ao Brasil, Cuba e Argentina em ocasiões diferentes.Â
Já a análise filogeográfica do subgenĂłtipo D2 sugeriu que o mais provável epicentro tenha sido a Europa Central e Oriental, com destaque para a RĂşssia, incluindo paĂses como EstĂ´nia, PolĂ´nia e SĂ©rvia. As sequĂŞncias genĂ©ticas dos Estados Unidos, quando agrupadas com as indianas, apontaram que pelo menos duas introduções diferentes deste subgenĂłtipo ocorreram no paĂs norte-americano a partir da ĂŤndia. O principal perĂodo de introdução foi estimado entre 1966 e 1978.Â
O subgenĂłtipo D3 apresentou vias de dispersĂŁo complexas, incluindo diferentes regiões geográficas e mĂşltiplas introduções. As análises destacaram que o D3 tenha como origem provável o Sul da Europa (Itália e Espanha) e o Brasil como fonte de dispersĂŁo para o continente. O subgenĂłtipo D3 foi encontrado em todas as regiões do Brasil, com as mais altas taxas na RegiĂŁo Sul, que recebeu um fluxo intenso de imigrantes europeus.Â
A investigação do D4 demonstrou uma maior prevalĂŞncia desse subgenĂłtipo em paĂses americanos onde a população majoritária Ă© de descendentes de africanos. Entretanto, os resultados sobre o local de origem do D4 foram inconclusivos. “Somente nĂŁo foi possĂvel desenhar a árvore filogenĂ©tica do subgenĂłtipo D7, devido Ă sua natureza recombinante e ao nĂşmero limitado de sequĂŞncias disponĂveis”, ponderou a especialista.
Os pesquisadores destacaram que, de forma majoritária, os achados foram compatĂveis com dados histĂłricos e epidemiolĂłgicos, demonstrando assim a utilidade das ferramentas de bioinformática em estudos evolutivos. A migração europeia em massa para as AmĂ©ricas ocorreu do inĂcio do sĂ©culo XIX atĂ© meados do sĂ©culo XX, especialmente para os Estados Unidos, Argentina, Canadá, Brasil, Cuba e Uruguai. Em perĂodo semelhante, milhares de pessoas, principalmente da SĂria, LĂbano, Palestina, Turquia e Egito migraram para Cuba. Em relação ao Brasil, o movimento migratĂłrio de árabes, em especial de paĂses como SĂria e LĂbano, tambĂ©m Ă© documentado a partir da segunda metade do sĂ©culo XIX. Já nos Estados Unidos, um dos maiores grupos de imigrantes tem como origem a ĂŤndia.
O estudo foi realizado como parte do projeto de doutorado da estudante Natália Spitz Toledo Dias, do Programa de PĂłs-graduação Stricto sensu em Biologia Parasitária do IOC. “As análises tambĂ©m permitiram conhecer melhor a velocidade com que o vĂrus evolui e a forma como ele vai se modificando ao longo do tempo”, frisou. “Determinadas mutações sĂŁo importantes do ponto de vista clĂnico, uma vez que podem gerar resistĂŞncia a um medicamento antiviral, falha na resposta Ă vacina ou fazer com que o microrganismo nĂŁo seja reconhecido nos kits de diagnĂłstico, por exemplo”, completou.Â
A pesquisa, publicada no periĂłdico cientĂfico Plos One, tambĂ©m contou com a contribuição dos pesquisadores do IOC Gonzalo Bello, do LaboratĂłrio de Aids e Imunologia Molecular; Selma Gomes, do LaboratĂłrio de Virologia Molecular; Francisco Mello, do LaboratĂłrio de Hepatites Virais, e Aline Moreira, do LaboratĂłrio de GenĂ´mica Funcional e Bioinformática. Participaram, ainda, especialistas da Universidade Federal de Goiás (UFG) e da Fundação Estadual de Produção e Pesquisa em SaĂşde (FEPPS), do Rio Grande do Sul.
Permitida a reprodução sem fins lucrativos do texto desde que citada a fonte (Comunicação / Instituto Oswaldo Cruz)