Cientistas da Fundação Oswaldo Cruz (Fiocruz) lideram um esforço nacional de pesquisa que busca decodificar o genoma do SARS-CoV-2, causador da Covid-19, e acompanhar suas linhagens e mutações genĂ©ticas. Por meio da Rede GenĂ´mica Fiocruz, especialistas de todas as unidades da Fundação no paĂs e de institutos parceiros se empenham diariamente em gerar dados mais robustos sobre o comportamento do vĂrus e contribuir para um melhor preparo do paĂs no enfrentamento da pandemia em termos de diagnĂłstico mais precisos e vacinas eficazes.
O grupo participa da iniciativa internacional de acesso aberto a informações sobre genomas de vĂrus influenza e coronavĂrus, o GISAID. Desde o inĂcio da pandemia, mais de 524 mil sequĂŞncias genĂ´micas do novo coronavĂrus já foram compartilhadas por pesquisadores de todo o mundo.
Com foco na situação do Brasil, a Rede GenĂ´mica Fiocruz passou a disponibilizar dados exclusivos do paĂs no website da iniciativa. “Para o rápido progresso no entendimento da Covid-19 e suas complicações Ă© fundamental estreitar a colaboração cientĂfica. Por meio dos dados genĂ´micos Ă© possĂvel avançar em pesquisa e desenvolvimento de possĂveis vacinas e medicamentos, alĂ©m de manter os kits de diagnĂłstico sempre atualizados com os genomas circulantes e a vigilância da dispersĂŁo dos vĂrus sempre alerta, ainda mais em um momento em que está sendo observado o surgimento de variantes do novo coronavĂrus", frisa a chefe do LaboratĂłrio de VĂrus RespiratĂłrios e do Sarampo do Instituto Oswaldo Cruz (IOC/Fiocruz), Marilda Siqueira, que atua como Centro de ReferĂŞncia Nacional em vĂrus respiratĂłrios junto ao MinistĂ©rio da SaĂşde e como referĂŞncia para a Organização Mundial da SaĂşde em Covid-19 nas AmĂ©ricas.
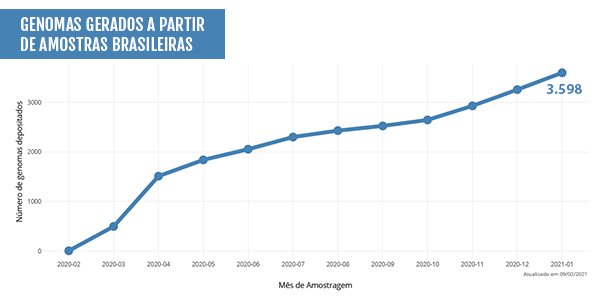
De acordo com os dados disponĂveis no site da Rede GenĂ´mica Fiocruz, já foram realizados cerca de 3,6 mil sequenciamentos do genoma do SARS-CoV-2 no Brasil, sendo SĂŁo Paulo o estado com o maior nĂşmero de amostras sequenciadas, com um total de 1.035, seguido por Rio de Janeiro, com 726, Amazonas, 340, Rio Grande do Sul, 306, ParaĂba, 167, e Pernambuco, com 150.
“Quanto maior for o investimento na ciência, mais rápidas serão as respostas ao atual e a futuros problemas de saúde pública”, salienta a coordenadora da curadoria da plataforma genômica GISAID no Brasil, a pesquisadora Paola Cristina Resende, do mesmo Laboratório.
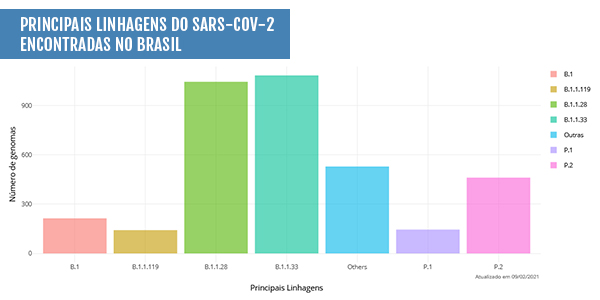
Segundo a iniciativa, mais de 60 linhagens do novo coronavĂrus já foram identificadas no paĂs. No entanto, os especialistas destacam o predomĂnio das linhagens B.1.1.33 e B.1.1.28, que apresentam circulação nacional desde março de 2020. A linhagem B.1.1.28 deu origem a duas linhagens que passaram a circular mais recentemente no paĂs, a P.1 e a P.2, denominadas cientificamente de “variantes de preocupação”, ou variant of concern (VOC), na expressĂŁo em inglĂŞs.
“A identificação da circulação de linhagens nĂŁo tem ligação direta com gravidade da doença, aumento da transmissibilidade do patĂłgeno ou impacto na eficácia das vacinas. No caso dos vĂrus, por exemplo, a maioria das mutações nĂŁo causa mudanças na capacidade de dispersĂŁo ou infecção. O SARS-CoV-2 muda mais rapidamente que outros microrganismos, como bactĂ©rias e fungos. Na maior parte dos casos, as linhagens sĂŁo distintas por pequenas diferenças em seu material genĂ©tico, que podem nĂŁo ser associadas a novas caracterĂsticas virais”, explica Marilda.
No estado mais populoso do paĂs, SĂŁo Paulo, as variantes B.1.1.28 e B.1.1.33 sĂŁo, atĂ© o momento, as que apresentam maior nĂşmero de identificação. Já no Rio de Janeiro, a B.1.1.33 apresentou maior prevalĂŞncia por um perĂodo. No entanto, nos Ăşltimos meses, a cepa P.2 tem demonstrado crescimento no estado, assim como em todo o paĂs.
Em relação ao Amazonas, que enfrenta sĂ©rios problemas em seu sistema de saĂşde, os dados indicam o avanço acelerado da linhagem P.1. A recente variante, derivada de uma das linhagens que predominava no paĂs, a B.1.1.28, Ă© responsável atualmente por mais de 91% das infecções no estado. Estudos indicam que a variante contĂ©m pelo menos trĂŞs mutações (K417N, E484K e N501Y), que afetam a proteĂna Spike, associada Ă entrada do vĂrus nas cĂ©lulas humanas. A P.1 tambĂ©m foi identificada no Pará, Roraima, ParaĂba, SĂŁo Paulo e Santa Catarina. Confira o artigo sobre a relação filogenĂ©tica de sequĂŞncias de SARS-CoV-2 do Amazonas com as variantes emergentes brasileiras que abrigam mutações.
“Com o objetivo de detectar a circulação desta linhagem entre a população do Amazonas e outros estados do paĂs, estamos recebendo de 30 a 40 amostras positivas de diferentes localidades para realizarmos o sequenciamento e entendermos a possĂvel dispersĂŁo dessa e de outras cepas”, explica Fernando Motta, pesquisador do mesmo LaboratĂłrio. “EstĂŁo sendo conduzidos estudos com amostras isoladas das diferentes linhagens a fim de identificar como as mutações recĂ©m descritas podem afetar ou nĂŁo a reposta induzida pelas vacinas em uso”, completa o virologista.
“É importante lembrar que as linhagens P.1 e P.2 já foram associadas a casos de reinfecção no paĂs. Por isso, Ă© fundamental a continuidade das medidas de prevenção, como a utilização de máscara de proteção, a higienização frequente das mĂŁos e evitar aglomerações”, ressalta Paola. Confira detalhes aqui e aqui.
“Assim como há dĂ©cadas existe uma rede de vigilância nacional muito bem estruturada para acompanhamento dos vĂrus influenza, causadores da gripe, que circulam em todo o mundo e sofrem mutações frequentes, nossa rede permanecerá igualmente empenhada nas vigilâncias epidemiolĂłgica e laboratorial do novo coronavĂrus, fundamentais para a formulação de vacinas sempre atualizadas e eficazes, por exemplo”, ressalta Marilda.
A Rede GenĂ´mica Fiocruz integra pesquisadores de todas as unidades da Fundação no paĂs: Instituto Aggeu MagalhĂŁes (Fiocruz-PE), Instituto Gonçalo Moniz (Fiocruz-BA), Instituto RenĂ© Rachou (Fiocruz-MG), Instituto Carlos Chagas (Fiocruz-PR), Instituto Leonidas e Maria Deane (Fiocruz-AM), assim como as unidades no Mato Grosso do Sul, RondĂ´nia, PiauĂ e Ceará. TambĂ©m participam especialistas do Instituto Oswaldo Cruz (IOC/Fiocruz), Instituto Nacional de Controle de Qualidade em SaĂşde (INCQS/Fiocruz), Instituto Nacional de SaĂşde da Mulher, da Criança e do Adolescente Fernandes Figueira (IFF/Fiocruz), alĂ©m do Instituto Adolfo Lutz, de SĂŁo Paulo, e Instituto Evandro Chagas, do Pará.
Cientistas da Fundação Oswaldo Cruz (Fiocruz) lideram um esforço nacional de pesquisa que busca decodificar o genoma do SARS-CoV-2, causador da Covid-19, e acompanhar suas linhagens e mutações genĂ©ticas. Por meio da Rede GenĂ´mica Fiocruz, especialistas de todas as unidades da Fundação no paĂs e de institutos parceiros se empenham diariamente em gerar dados mais robustos sobre o comportamento do vĂrus e contribuir para um melhor preparo do paĂs no enfrentamento da pandemia em termos de diagnĂłstico mais precisos e vacinas eficazes.
O grupo participa da iniciativa internacional de acesso aberto a informações sobre genomas de vĂrus influenza e coronavĂrus, o GISAID. Desde o inĂcio da pandemia, mais de 524 mil sequĂŞncias genĂ´micas do novo coronavĂrus já foram compartilhadas por pesquisadores de todo o mundo.
Pesquisadores já conseguiram sequenciar quase 3,6 mil amostras brasileiras
Com foco na situação do Brasil, a Rede GenĂ´mica Fiocruz passou a disponibilizar dados exclusivos do paĂs no website da iniciativa. “Para o rápido progresso no entendimento da Covid-19 e suas complicações Ă© fundamental estreitar a colaboração cientĂfica. Por meio dos dados genĂ´micos Ă© possĂvel avançar em pesquisa e desenvolvimento de possĂveis vacinas e medicamentos, alĂ©m de manter os kits de diagnĂłstico sempre atualizados com os genomas circulantes e a vigilância da dispersĂŁo dos vĂrus sempre alerta, ainda mais em um momento em que está sendo observado o surgimento de variantes do novo coronavĂrus", frisa a chefe do LaboratĂłrio de VĂrus RespiratĂłrios e do Sarampo do Instituto Oswaldo Cruz (IOC/Fiocruz), Marilda Siqueira, que atua como Centro de ReferĂŞncia Nacional em vĂrus respiratĂłrios junto ao MinistĂ©rio da SaĂşde e como referĂŞncia para a Organização Mundial da SaĂşde em Covid-19 nas AmĂ©ricas.
De acordo com os dados disponĂveis no site da Rede GenĂ´mica Fiocruz, já foram realizados cerca de 3,6 mil sequenciamentos do genoma do SARS-CoV-2 no Brasil, sendo SĂŁo Paulo o estado com o maior nĂşmero de amostras sequenciadas, com um total de 1.035, seguido por Rio de Janeiro, com 726, Amazonas, 340, Rio Grande do Sul, 306, ParaĂba, 167, e Pernambuco, com 150.
“Quanto maior for o investimento na ciência, mais rápidas serão as respostas ao atual e a futuros problemas de saúde pública”, salienta a coordenadora da curadoria da plataforma genômica GISAID no Brasil, a pesquisadora Paola Cristina Resende, do mesmo Laboratório.
Segundo a iniciativa, mais de 60 linhagens do novo coronavĂrus já foram identificadas no paĂs. No entanto, os especialistas destacam o predomĂnio das linhagens B.1.1.33 e B.1.1.28, que apresentam circulação nacional desde março de 2020. A linhagem B.1.1.28 deu origem a duas linhagens que passaram a circular mais recentemente no paĂs, a P.1 e a P.2, denominadas cientificamente de “variantes de preocupação”, ou variant of concern (VOC), na expressĂŁo em inglĂŞs.
Â
O sequenciamento permite identificar todas as caracterĂsticas genĂ©ticas do vĂrus, conhecimento fundamental para a formulação de vacinas e fármacos
“A identificação da circulação de linhagens nĂŁo tem ligação direta com gravidade da doença, aumento da transmissibilidade do patĂłgeno ou impacto na eficácia das vacinas. No caso dos vĂrus, por exemplo, a maioria das mutações nĂŁo causa mudanças na capacidade de dispersĂŁo ou infecção. O SARS-CoV-2 muda mais rapidamente que outros microrganismos, como bactĂ©rias e fungos. Na maior parte dos casos, as linhagens sĂŁo distintas por pequenas diferenças em seu material genĂ©tico, que podem nĂŁo ser associadas a novas caracterĂsticas virais”, explica Marilda.
No estado mais populoso do paĂs, SĂŁo Paulo, as variantes B.1.1.28 e B.1.1.33 sĂŁo, atĂ© o momento, as que apresentam maior nĂşmero de identificação. Já no Rio de Janeiro, a B.1.1.33 apresentou maior prevalĂŞncia por um perĂodo. No entanto, nos Ăşltimos meses, a cepa P.2 tem demonstrado crescimento no estado, assim como em todo o paĂs.
Em relação ao Amazonas, que enfrenta sĂ©rios problemas em seu sistema de saĂşde, os dados indicam o avanço acelerado da linhagem P.1. A recente variante, derivada de uma das linhagens que predominava no paĂs, a B.1.1.28, Ă© responsável atualmente por mais de 91% das infecções no estado. Estudos indicam que a variante contĂ©m pelo menos trĂŞs mutações (K417N, E484K e N501Y), que afetam a proteĂna Spike, associada Ă entrada do vĂrus nas cĂ©lulas humanas. A P.1 tambĂ©m foi identificada no Pará, Roraima, ParaĂba, SĂŁo Paulo e Santa Catarina. Confira o artigo sobre a relação filogenĂ©tica de sequĂŞncias de SARS-CoV-2 do Amazonas com as variantes emergentes brasileiras que abrigam mutações.
“Com o objetivo de detectar a circulação desta linhagem entre a população do Amazonas e outros estados do paĂs, estamos recebendo de 30 a 40 amostras positivas de diferentes localidades para realizarmos o sequenciamento e entendermos a possĂvel dispersĂŁo dessa e de outras cepas”, explica Fernando Motta, pesquisador do mesmo LaboratĂłrio. “EstĂŁo sendo conduzidos estudos com amostras isoladas das diferentes linhagens a fim de identificar como as mutações recĂ©m descritas podem afetar ou nĂŁo a reposta induzida pelas vacinas em uso”, completa o virologista.
“É importante lembrar que as linhagens P.1 e P.2 já foram associadas a casos de reinfecção no paĂs. Por isso, Ă© fundamental a continuidade das medidas de prevenção, como a utilização de máscara de proteção, a higienização frequente das mĂŁos e evitar aglomerações”, ressalta Paola. Confira detalhes aqui e aqui.
“Assim como há dĂ©cadas existe uma rede de vigilância nacional muito bem estruturada para acompanhamento dos vĂrus influenza, causadores da gripe, que circulam em todo o mundo e sofrem mutações frequentes, nossa rede permanecerá igualmente empenhada nas vigilâncias epidemiolĂłgica e laboratorial do novo coronavĂrus, fundamentais para a formulação de vacinas sempre atualizadas e eficazes, por exemplo”, ressalta Marilda.
A Rede GenĂ´mica Fiocruz integra pesquisadores de todas as unidades da Fundação no paĂs: Instituto Aggeu MagalhĂŁes (Fiocruz-PE), Instituto Gonçalo Moniz (Fiocruz-BA), Instituto RenĂ© Rachou (Fiocruz-MG), Instituto Carlos Chagas (Fiocruz-PR), Instituto Leonidas e Maria Deane (Fiocruz-AM), assim como as unidades no Mato Grosso do Sul, RondĂ´nia, PiauĂ e Ceará. TambĂ©m participam especialistas do Instituto Oswaldo Cruz (IOC/Fiocruz), Instituto Nacional de Controle de Qualidade em SaĂşde (INCQS/Fiocruz), Instituto Nacional de SaĂşde da Mulher, da Criança e do Adolescente Fernandes Figueira (IFF/Fiocruz), alĂ©m do Instituto Adolfo Lutz, de SĂŁo Paulo, e Instituto Evandro Chagas, do Pará.
Permitida a reprodução sem fins lucrativos do texto desde que citada a fonte (Comunicação / Instituto Oswaldo Cruz)